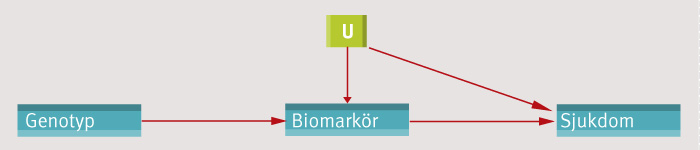
I denna figur representerar U alla förväxlingsfaktorer som kan tänkas påverka både biomarkören och sjukdomen. Om det inte finns några förväxlingsfaktorer kan det ojusterade statistiska sambandet mellan biomarkör och sjukdom ges en kausal tolkning, vilket gör den mer sofistikerade mendelska randomiseringsanalysen onödig. (Grafik: LT)
Peter M Nilsson beskriver i LT 16/2013 den epidemiologiska analysmetoden mendelsk randomisering (MR). Eftersom mendelsk randomisering används allt oftare (se till exempel referenserna i Nilssons artikel) är det viktigt att tydliggöra metodens förutsättningar och antaganden, och jag vill därför korrigera Nilsson på en central punkt.
I bildtexten till Figur 1 skriver Nilsson »Principen för mendelsk randomisering … utgår från att en genetisk markör inte bara har ett samband med en förmedlande biomarkör … utan den genetiska markören ska också kunna uppvisa ett eget oberoende samband med sjukdomen i fråga«, vilket illustreras av en direkt pil från genotyp till sjukdom i Nilssons figur.
Detta är felaktigt, principen för mendelsk randomisering är tvärtom att genotypen inte ska uppvisa ett oberoende samband med sjukdomen. Mer precist: genotypen ska inte ha en direkt effekt på sjukdomen, utan all effekt ska vara medierad genom biomarkören (Nilsson påpekar visserligen detta senare i Fakta 1, men detta påpekande motsägs av Figur 1). Det rätta antagandet illustreras av frånvaron av en direkt pil från genotyp till sjukdom i min figur. I denna figur representerar U alla faktorer som kan tänkas påverka både biomarkören och sjukdomen, så kallade förväxlingsfaktorer (confounders). Om det inte finns några förväxlingsfaktorer kan det ojusterade statistiska sambandet mellan biomarkör och sjukdom ges en kausal tolkning, vilket gör den mer sofistikerade mendelska randomiseringsanalysen onödig.
Att frånvaron av en direkt effekt av genotypen är avgörande för giltigheten av mendelsk randomiseringsanalys kan inses på följande sätt. Den enklaste varianten av mendelsk randomiseringsanalys innebär att vi studerar det statistiska sambandet mellan genotyp och sjukdom, och ignorerar biomarkören i analysen. Om vi observerar ett samband antas detta bevisa att biomarkören har en kausal effekt på sjukdomen. Detta bevis är giltigt under förutsättning att en direkt effekt av genotypen saknas, eftersom sambandet i så fall bara kan förklaras av den kausala kedjan genotyp→biomarkör→sjukdom. Har genotypen däremot en direkt effekt (som i Nilssons figur) kan ju denna också förklara det observerade sambandet, och den mendelska randomiseringsanalysen saknar därmed bevisvärde.
En viktig fråga är huruvida frånvaron/närvaron av en direkt effekt kan undersökas empiriskt. En vanlig (miss)uppfattning (se till exempel referens 11 i Nilssons artikel) är att detta kan göras genom att testa huruvida det finns ett statistiskt samband mellan genotypen och sjukdomen, givet (egentligen inom strata av) biomarkören.
Det finns två problem med detta test:
1) Om det finns förväxlingsfaktorer (U) är det fullt möjligt att genotypen och sjukdomen uppvisar ett statistiskt samband, givet biomarkören, även om genotypen inte har någon direkt effekt på sjukdomen [1]. Testet är alltså inte giltigt om det finns förväxlingsfaktorer.
2) Ett statistiskt test kan leda till att »nollhypotesen« (det vill säga hypotesen om statistiskt oberoende) förkastas, men kan inte leda till att nollhypotesen accepteras; att p-värdet är större än testets signifikansnivå (0,05) indikerar inte att nollhypotesen är sann [2].
Vi kan alltså använda testet för att indikera att en viss genotyp har en direkt effekt på sjukdomen (i frånvaro av förväxlingsfaktorer) och därmed är olämplig för mendelsk randomiseringsanalys, men vi kan inte använda testet för att indikera att en viss genotyp saknar direkt effekt på sjukdomen (oavsett om förväxlingsfaktorer är frånvarande eller närvarande) och därmed är lämplig för mendelsk randomiseringsanalys.
En utförlig diskussion av den mendelska randomiseringsanalysens antaganden, och i vilken utsträckning dessa är testbara, ges av Glymour et al [3]. Författarna konkluderar: »It is not generally possible to prove MR assumptions, but it is often possible to find empirical (though usually not conclusive) evidence suggesting that the putative IV* is invalid.«
*IV, instrumental variable, det vill säga genotyp i den mendelska randomiseringsanalysen.
Läs repliken: